遗传算法库
4.93/5 (174投票s)
遗传算法的框架
遗传算法库概述
遗传算法
遗传算法在一组可能的解上进行操作。由于遗传算法的随机性,算法找到的解可能是好的、差的,或不可行的[有缺陷的、错误的],因此应该有一种方法来指定该解的好坏程度。这是通过为解分配适应度值[或仅是适应度]来实现的。染色体在遗传算法中表示解。染色体的两个基本组成部分是编码的解及其适应度值。
染色体被分组到种群[一组解]中,遗传算法在此种群上进行操作。在每个步骤[代]中,遗传算法从种群中选择染色体[选择通常基于染色体的适应度值]并组合它们以产生新的染色体[后代]。这些后代染色体形成一个新的种群[或替换现有种群中的一些染色体],希望新的种群会比之前的种群更好。种群会跟踪最差和最好的染色体,并存储可供遗传算法用于确定停止条件的附加统计信息。
染色体以某种方式存储其所代表的解。这称为解的表示[编码]。有许多可能的方式来表示一个解,使其适合遗传算法[二进制、实数、实数向量、排列等],它们大多取决于问题的性质。


遗传算法通过组合现有染色体来产生新的染色体[解]。这个操作称为交叉。交叉操作从两个现有染色体[父代]中获取解编码的一部分,并将它们组合成一个单一的解[新染色体]。这个操作取决于染色体表示,并且可能非常复杂。虽然通用的交叉操作易于实现,但为特定问题构建专门的交叉操作可以极大地提高遗传算法的性能。

在遗传算法完成新染色体的生成之前,在执行交叉操作之后,它会执行变异操作。变异操作对编码的解进行随机但微小的更改。这可以防止所有解陷入局部最优,并扩展算法的搜索空间。变异和交叉操作都取决于所选的表示。

在产生新染色体时,交叉和变异操作并非总是执行。如果不执行交叉,遗传算法将通过复制其中一个父代来产生新染色体。交叉和变异操作的速率分别称为交叉概率和变异概率。交叉概率通常很高[约 80%],而变异概率应该相对较低[约 3%,但对于某些问题,较高的概率会产生更好的结果]。较高的变异概率可能将遗传算法变成随机搜索算法。
遗传算法用于操纵染色体的最后两个操作是适应度操作和适应度比较器。适应度操作衡量所产生解[染色体]的质量。这个操作特定于问题,并且它实际上告诉遗传算法要优化什么。适应度比较器[顾名思义]用于基于其适应度比较染色体。基本上,适应度比较器告诉遗传算法是应该最小化还是最大化染色体的适应度值。
从种群中为产生新染色体选择父代称为选择。选择可以基于许多不同的标准,但通常基于适应度值。其背后的想法是选择最好的染色体作为父代,希望组合它们会产生更好的后代染色体。但是,只选择最好的染色体有一个主要的缺点,种群中的所有染色体都会很快变得相同。这缩小了探索空间,将遗传算法推入局部最优,并阻止遗传算法找到可能存在的、位于探索空间中难以访问的区域的更好解。为了保持种群中染色体的多样性[以及更广阔的探索空间],选择操作通常会在选择过程中引入随机因素。一些选择操作的实现是完全随机的。
基于适应度值的选择操作可能会出现一个问题。当存在具有主导适应度值的染色体时,它会被 most of the times 选中,从而导致现有类似的问题。为防止这种情况,可以对适应度值进行缩放[转换]以减小主导染色体与其他染色体之间的差异[这允许选择其他染色体]。转换适应度值的方法有很多。通常,它们通过对适应度值应用数学转换来实现,但还有其他方法,如基于排名的缩放,使用染色体的排名[基于染色体的原始适应度值]作为缩放后的适应度值。

耦合操作定义了如何将选定的染色体[父代]配对进行交配[交配是通过对配对的父代执行交叉操作并对新产生的染色体应用变异操作来完成的]。此操作对产生新染色体提供了更好的控制,但也可以跳过它,并且可以在选择操作从种群中选择父代时产生新染色体。


遗传算法执行的下一步是向种群引入新染色体。后代染色体可以形成一个新的种群并替换整个[之前的]种群[非重叠种群],或者它们只能替换当前种群中的一些染色体[重叠种群]。对于重叠种群,替换操作定义了哪些染色体被移除[通常是最差的染色体]以及哪些后代染色体被插入。通过替换染色体,遗传算法有可能丢失[迄今为止]找到的最佳染色体。为防止这种情况,在遗传算法中引入了精英主义的概念。精英主义确保当前一代中的最佳染色体将存活到下一代。
算法逐个按顺序执行前面描述的步骤,当它们被执行完毕后,就说一代已经过去。在每一代结束时,遗传算法会检查停止条件。由于遗传算法的性质,大多数时候,不清楚算法何时应该停止,因此标准通常基于统计信息,例如代数、最佳染色体的适应度值、种群中染色体的平均适应度值、进化过程的持续时间……

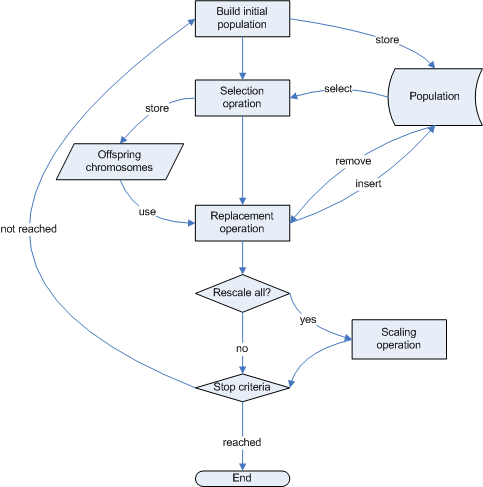

遗传算法库
这是对遗传算法库的设计和结构的简要介绍。该库是一组 C++ 类,它们代表了遗传算法的构建模块。
注意:有关库近期版本更改的更多详细信息,请参阅文章的此部分。
库的结构
下图说明了遗传算法库的基本结构

第一层主要包含与遗传算法不直接相关但对其实现至关重要的类。遗传算法库实现了随机数生成器、一组用于平台无关的线程和同步的类、用于更轻松地管理内存的智能指针[主要用于自动管理染色体使用的内存],以及目录[目录用于存储和跟踪当前可用的遗传操作]。
除了这些通用功能外,库还在最底层提供了一些更特定于 GA 的东西,例如用于跟踪遗传算法统计信息和观察框架的类。遗传操作和参数对象的接口也在此层定义。
总之,这些功能提供了库其他更高级别所使用的通用功能。此层的类被划分为多个命名空间。
中层被划分为三个命名空间,如图所示。库的大部分核心功能在此层实现。
首先,Chromosome 命名空间包含一组接口和类,用于表示库中的染色体并定义它们在系统中的基本行为。此命名空间包含四种类型遗传操作的接口声明:交叉、变异、适应度操作和适应度比较器。
第二个命名空间是 Population 命名空间。此命名空间中的核心类是一个管理染色体种群、存储统计信息以及跟踪最佳和最差染色体的类。选择、耦合、替换和缩放操作的接口在此命名空间中定义。
最后一个命名空间 Algorithm 定义了遗传算法的接口,并实现了其一些基本行为。此命名空间还定义了一个实现算法停止条件的接口。
这两层代表了库的核心。
库的顶层实现了前面提到的遗传操作、染色体表示和遗传算法。如前所述,所有内置遗传操作都按类别排序。

染色体
染色体是遗传算法中的核心对象。染色体在此库中由 GaChromosome 类定义。

GaChromosome 类GaChromosome 是实际染色体实现的接口。此类[接口]定义了 Crossover、Mutation、CalculateFitness 和 CompareFitness 方法,它们代表了前面描述的遗传操作[变异、交叉、适应度操作和适应度比较器]。
MakeCopy 方法代表一个虚拟复制构造函数。新类应该重写此方法,它应该返回染色体对象的新实例。此方法可以复制整个染色体[设置和编码的解],或者只复制染色体的设置[保留空的编码]。MakeFromPrototype 方法创建一个具有与当前对象相同设置的新染色体对象,但它会随机初始化解的编码。
每个染色体都有定义的参数[如交叉和变异概率],这些参数由 GaChromosomeParams 类表示。

GaChromosomeParams 类GaChromosomeParams 定义了变异和交叉概率、变异大小以及交叉点数,以及“仅改进变异”标志。默认构造函数初始化的默认染色体参数为
- 变异概率:3%
- 变异大小:1[仅一个编码解的值被变异]
- 仅接受改进变异:是
- 交叉概率:80%
- 交叉点数:1
库中的所有参数类都继承自 GaParameters 类。
![]()
GaParameters 类此类[接口]定义了 Clone 方法,它代表一个虚拟复制构造函数,并且应该返回指向参数对象副本的新对象指针。
GaChromosomes 类是一个接口,它不实现任何行为。尽管如此,所有染色体类型都具有一些共同的基本特征[存储染色体参数和适应度值],这些特征由 GaDefaulChromosome 类实现。除了参数,染色体还可以有其他配置数据[**C**hromosome **C**onfiguration **B**lock 或 CCB],并且这些数据通常对种群中的所有染色体都相同。染色体配置数据的存储由 GaChromosomeParamBlock 类定义。

GaDefaultChromosome 和 GaChromosomeParamsBlock 类GaDefaultChromosome 类的 Crossover 和 Mutation 方法仅以由染色体参数定义的概率执行这些遗传操作。如果应该执行这些操作,这些方法会调用 PerformCrossover 和 PerformMutation。继承 GaDefaultChromosome 类的新染色体应该重写 PerformCrossover 和 PerformMutation,而不是 Crossover 和 Mutation 方法。
此类还引入了一个用于改进变异的框架。在执行变异操作之前,会调用 PrepareForMutation 方法。此方法应备份旧染色体,然后执行变异。之后,比较染色体[变异前]的旧适应度和新适应度。如果变异提高了适应度,则接受它,并调用 AcceptMutation 方法;否则,调用 RejectMutation 方法。如果未设置“仅改进变异”标志,则变异会立即被接受,而无需调用这些方法。
可以通过继承已定义的实现特定染色体类型的类来实现交叉、变异和适应度操作。然后,用户可以重写 PerformCrossover [或 Crossover]、PerformMutation [或 Mutation] 和 CalculateFitness 方法,并为目标问题实现特定的操作。
遗传算法库提供了另一种实现此目的的方法。这些遗传操作可以在独立的类中定义和实现。然后,可以将这些类的对象引用[称为仿函数]存储在 CCB 中。这允许用户在运行时更改遗传操作[这是通过前一种方法无法实现的]。

GaDynamicOperationChromosome 类和遗传操作接口GaDynamicOperationChromosome 重写了 PerformCrossover、PerformMutation、CalculateFitness 和 CompareFitnesses 方法,并将调用路由到存储在 CCB 中的遗传操作仿函数。
由 GaChromosomeOperationsBlock 类表示的 CCB 存储这些仿函数。
GaCrossoverOperation 是交叉操作的接口。用户定义的类应该重写 operator()
virtual GaChromosomePtr operator ()(
const GaChromosome* parent1,
const GaChromosome* parent2) const;
参数是指由交叉操作使用的父代指针。该运算符应返回子代染色体的智能指针。
GaMutationOperation 是变异操作的接口。用户定义的类应该重写 operator()
virtual void operator ()(GaChromosome* chromosome) const;
此运算符[作为参数]接受一个指向应执行此操作的染色体的指针。
GaFitnessOperation 是适应度操作的接口。用户定义的类应该重写 operator()
virtual float operator ()(const GaChromosome* chromosome) const;
此运算符[作为参数]接受一个指向应执行此操作的染色体的指针,并返回染色体的计算适应度值。
最后一个操作是适应度比较器。适应度比较器的接口由 GaFitnessComparator 类定义。用户定义的类应该重写 operator()
virtual int operator ()
float fitness1,
float fitness2) const;
此运算符接受两个适应度值作为参数,并返回一个整数
- -1 如果第一个适应度值低于第二个
- 0 如果这两个值相等
- 1 如果第一个值高于第二个
实现库中遗传操作的所有类都继承自 GaOperation 类。

GaOperation 类MakeParameters 创建操作所需的参数对象,并返回指向该对象的指针。如果操作不需要参数,则该方法可以返回 NULL 指针。CheckParameters 方法检查提供的参数的有效性,如果参数有效则返回 true,如果无效则返回 false。所有遗传操作都必须实现这两个方法。
遗传算法库被设计为使用无状态的遗传操作对象[仿函数]。遵循此设计,所有内置操作都是无状态的,但库可以处理对象不是无状态的用户定义操作。
种群
遗传算法的种群对象在此库中由 GaPopulation 类表示。

GaPopulation 类种群对象存储染色体和统计信息。种群中的染色体由 GaScaledChromosome 类的对象表示。此类对象将染色体绑定到种群。染色体通常存储不依赖于其所在种群的数据,但染色体有一部分信息是依赖于种群的[例如缩放后的适应度,或染色体在种群中的索引]。这些数据是 GaScaledChromosome 类的成员。这使得在种群之间共享染色体更加容易和[内存]高效。
染色体可以按排序或未排序的顺序[按适应度值 - 缩放的或原始的]存储在种群中。种群是否应该排序取决于遗传算法的其他部分[例如选择操作],并由种群的参数控制。当种群排序时,跟踪最佳和最差染色体也更容易,但耗时更长。如果未排序,种群会使用排序组[GaSortedGroup 类]来跟踪这些染色体。排序组存储种群中染色体的索引。要跟踪的染色体数量[在最佳和最差组中]由种群的参数定义。需要注意的是,跟踪大量染色体效率不高;在这种情况下,最好使用排序种群。
创建种群时,它不包含任何染色体[未初始化]。Initialize 方法通过使用提供的原型染色体的 MakeFromPrototype 方法创建新染色体来填充种群。

GaStatistics 类用于存储和跟踪种群的统计信息。此类对象存储当前代和上一代种群的信息。

GaStatistics 和支持类GaStatValue 模板类存储单个统计值。GaStatValueType 枚举定义了跟踪的统计数据。这些数据可用于衡量算法的进展,并且通常由停止条件使用。
种群的行为由种群的参数控制。GaPopulationParameters 类管理种群的参数。
参数只是种群配置的一部分。配置还包括遗传操作[在种群上执行的操作 - 选择、耦合、替换和缩放]及其参数。GaPopulationConfiguration 类存储和管理配置。配置可以在种群之间共享。BindPopulation 方法应用配置并将种群绑定到它。UnbindPopulation 指示种群不再使用该配置,并解除绑定种群。当配置的某个方面发生更改时,所有绑定的种群都会收到通知。
当创建种群配置的对象并使用默认构造函数进行初始化时,构造函数会复制默认配置。可以使用 GetDefault 方法获取默认配置的引用。用户可以在运行时更改默认配置。开始时,默认配置被初始化为
- 种群参数
- 种群大小:10
- 可调整大小:否
- 已排序:是
- 用于排序的缩放适应度值:否
- 跟踪最佳染色体:0 [排序种群]
- 跟踪最差染色体:0 [排序种群]
- 适应度比较器:
GaMaxFitnessComparator - 选择:
GaSelectRouletteWheel,大小:2 - 耦合:
GaInverseCoupling,大小:2 - 替换:
GaReplaceWorst,大小:2 - 缩放:无

遗传操作及其参数作为一对存储在配置中。该配置使用 GaOperationParametersPair 类来存储这些对。对对象存储指向操作对象的指针以及操作参数对象副本。

GaOperationParametersPair 类选择操作的接口由 GaSelectionOperation 类定义。用户定义的类应重写 operator()
virtual void operator ()(
const GaPopulation& population,
const GaSelectionParams& parameters,
GaSelectionResultSet& result) const;
选择操作接收种群对象的引用和选择参数的引用。它还接收用于存储所选染色体的结果集引用。结果集存储所选染色体在种群中的索引,按适应度值排序。GaSelectionResultSet 类封装了 GaSortdeGroup 类。GaSelectionOperation 类有一个 MakeResultSet 方法,该方法创建一个新的结果集实例并返回对其的引用。如果选择操作需要不同类型的结果集,用户定义的类可以重写此方法。
GaSelectionParams 是选择操作参数的基类。此类仅定义一个参数[对所有选择操作都通用],即选择大小。

耦合操作的接口由 GaCouplingOperation 类定义。用户定义的类应重写 operator ()
virtual void GACALL operator ()(
const GaPopulation& population,
GaCouplingResultSet& output,
const GaCouplingParams& parameters,
int workerId,
int numberOfWorkers) const=0;
耦合操作接收种群对象的引用和耦合参数的引用。它还接收用于存储产生的后代染色体及其父代信息的 `GaCouplingResultSet` 类的结果集引用。
耦合操作可以由多个工作线程并行执行。框架提供了一定数量的线程来执行操作,以及执行当前操作调用的线程 ID。
GaCouplingParams 是耦合操作参数的基类。此类仅定义一个参数[对所有耦合操作都通用],即应产生的后代染色体数量。

替换操作的接口由 GaReplacementOperation 类定义。用户定义的类应重写 operator()
virtual void GACALL operator ()(
GaPopulation& population,
const GaReplacementParams& parameters,
const GaCouplingResultSet& newChromosomes) const;
替换操作接收种群对象的引用、替换参数的引用以及包含应插入种群的后代染色体的耦合操作结果集的引用。
GaReplacementParams 是替换操作参数的基类。此类仅定义一个参数[对所有替换操作都通用],即应替换的染色体数量。

缩放操作的接口由 GaScalingOperation 类定义。用户定义的类应重写 operator()、IsRankingBase 和 NeedRescaling 方法
virtual float operator ()(
const GaScaledChromosome& chromosome,
const GaPopulation& population,
const GaScalingParams& parameters) const;
virtual bool IsRankingBased() const;
virtual bool NeedRescaling(
const GaPopulation& population,
const GaScalingParams& parameters) const;
operator()接收种群对象的引用和缩放参数的引用。- 如果缩放操作的实现基于染色体的排名,
IsRankingBased应返回true。否则,它应返回false。 GaScalingParams是缩放操作参数的基类。
缩放可以基于从一代到下一代可能变化的值,或者可以使用在评估过程中较长时间内保持不变的值,或者根本不改变的值。缩放后的适应度值在将新染色体插入种群时计算,但种群的变化可能需要重新缩放种群中所有染色体的适应度值。NeedRescaling 方法由框架调用以检查是否需要重新缩放,这发生在每一代结束时。如果此方法返回 true,则框架会重新缩放种群中所有染色体的适应度值。

算法
遗传算法对象是将描述的构建块粘合在一起的。它定义并控制进化过程,并决定其结束。遗传算法的接口由 GaAlgorithm 类定义。

GaAlgorithm 类StartSolving、StopSolving 和 PauseSolving 方法允许用户控制进化过程,并且 GetState 方法可用于获取其当前状态。当用户在运行时更改算法的任何部分[遗传操作或其参数]时,应在进行任何更改之前调用 BeginParameterChange 方法。当用户完成更改后,用户必须调用 EndParameterChange 方法。
GaAlgorithmParams 类代表算法参数的基类。
遗传算法通过观察者模式通知系统其余部分进化过程中的变化。用户必须调用 SubscribeObserver 方法才能开始接收这些通知。当不再需要通知时,用户可以调用 UnsubscribeObserver 方法。传递给这两个方法的对象必须是继承自 GaObserver [或 GaObserverAdapter] 类的实例。

GaObserver 是遗传算法观察者的接口。此接口方法的实现实际上是事件处理程序。当遗传算法中发生事件时,算法会遍历已订阅观察者的列表,并调用处理该事件的适当观察者。
virtual void StatisticUpdate(
const GaStatistics& statistics,
const GaAlgorithm& algorithm);
|
在算法的统计信息已更改时通知观察者。此事件发生在每一代结束时。 |
void NewBestChromosome(
const GaChromosome& newChromosome,
const GaAlgorithm& algorithm);
|
当算法找到比先前找到的最佳染色体更好的新染色体时,发生此事件。 |
void EvolutionStateChanged(
GaAlgorithmState newState,
const GaAlgorithm& algorithm);
|
当用户启动、停止或暂停进化过程,或者当算法达到停止条件时,此事件通知观察者。 |
观察者列表由 GaObserverList 管理。此类还实现了 GaObserver 接口,但它不处理事件,而是将通知路由给所有已订阅的观察者。operator+= 和 operator-= 用于订阅和取消订阅观察者。
// subscribe observer
observerList += observer;
// ussubscribe observer
observerList -= observer;
当用户定义的观察者继承 GaObserver 类时,它必须实现所有定义的方法。有时,用户不想接收所有事件。在这种情况下,用户可以从观察者继承 GaObserverAdapter 类作为基类。GaObserverAdapter 类使用默认事件处理实现了 GaObserver 定义的所有方法;用户只能重写处理所需事件的方法。
进化过程的结束由停止条件决定。遗传算法库定义了 GaStopCriteria 类,它代表了这种遗传操作的接口。实现停止条件的自定义类应重写 Evaluate 方法
virtual bool Evaluate(
const GaAlgorithm& algorithm,
const GaStopCriteriaParams& parameters) const;
此方法接收对算法对象[其状态被评估]的引用以及停止条件参数的引用。如果算法已达到所需状态并且应停止进化,则此方法应返回 true。如果未达到条件,则该方法应返回 false。
GaStopCriteriaParams 是停止条件参数的基类。
遗传算法的一些默认行为在 GaBaseAlgorithm 类中实现。

GaBaseAlgorithm 类此类管理进化过程的状态和状态转换。下图说明了进化过程的可能状态、转换、触发转换的操作以及对状态更改的响应。

GaBaseAlgorithm 实现 StartSolving、StopSolving 和 PauseSolving 方法来控制进化过程。这些方法执行状态检查和状态转换。当进化过程的状态改变时,会调用以下虚拟方法之一:OnStart、OnResume、OnPause、OnStopt。实现特定遗传算法的新类应该重写这些方法来处理状态更改。
此类还管理已订阅观察者的列表,并通过实现 BeginParameterChange 和 EndParameterChange 方法来处理运行时更改,这些方法可以防止来自多个线程的并发更改。
遗传算法易于并行化,因为它们在独立解的集合上操作。这使得遗传算法能够以低的[实现和运行时]开销利用现代多处理器架构。
遗传算法库提供了一个遗传算法并行执行的框架。该框架围绕 GaMultithreadingAlgorithm 类构建。

GaMultithreadingAlgorithm 类每个多线程算法都有一个控制线程和至少一个工作线程[worker]。控制线程准备工作,然后将控制权转移给工作线程。在所有工作线程完成其工作部分后,控制线程再次获得控制权并合并工作线程产生的结果。此类管理所有使用的线程,因此用户不必担心多线程涉及的资源。
下图显示了多线程算法的控制流

GaMultithreadingAlgorithm 类重写了 OnStart、OnResume、OnPause 和 OnStop 方法来控制工作线程和控制线程。
ControlFlow 和 WorkFlow 方法代表了控制线程和工作线程的流程。这些方法提供了控制线程、工作线程和其余系统之间的同步和通信。在 ControlFlow 将控制权转移给工作线程之前,它会调用 BeforeWorkers 方法,在工作线程完成后,它会调用 AfterWorkers 方法。遗传算法实现可以重写这些方法来执行特定的工作准备和工作合并。当工作线程获得控制权时,WorkFlow 方法会调用 WorkStep 方法。此方法应用于执行所有可以并行执行的工作。
GaMultithreadingAlgorithmParams 是多线程算法参数的基类。它定义了在遗传算法执行中涉及的工作线程数量。
多线程
遗传算法库包含一组类、数据类型和宏,用于隔离平台相关的线程。GaThread 类封装和控制系统线程。

GaThread 类GaThreadStatus 枚举定义了线程的可能状态,GaThreadParameter 结构用于存储线程的启动参数。
库定义的线程数据类型
SystemThread |
此类型定义了用于存储线程对象的系统特定类型。 |
ThreadID |
此类型的变量/对象用于存储系统线程的 ID。此类型隐藏了用于此目的的系统特定类型。 |
ThreadFunctionPointer |
此类型定义了一个函数指针,用作线程的入口点。 |
ThreadFunctionReturn |
此类型用作用作线程入口点的函数的返回值类型。 |
GaCriticalSection 隔离了临界区免受多个线程并发访问的系统特定保护。GaSectionLock 类用于自动锁定和解锁临界区对象。
// ...
GaCriticalSection _criticalSection;
// ...
void SomeMethod()
{
// automatically acquires lock on _criticalSection synchronization object
GaSectionLock lock( &_criticalSection, true );
// ...critical section code...
// lock object is destroyed when execution leave this scope
// destructor of the object releases lock on used critical section object
}

GaCriticalSection 和 GaSectionLock 类遗传算法库定义了宏,可以更轻松地与临界区对象进行同步。这些宏稍后描述。
同步数据类型
SysSyncObject |
此类型定义了 GaCriticalSection 类使用的系统特定同步对象。 |
SysSemaphoreObject |
此类型定义了系统特定的信号量对象。 |
SysEventObject |
此类型定义了系统特定的事件对象。 |
同步函数
bool MakeSemaphore(
SysSemaphoreObject& semaphore,
int maxCount,
int initialCount);
|
此函数用于创建信号量的操作系统对象并进行初始化。 |
bool DeleteSemaphore(
SysSemaphoreObject& semaphore);
|
此函数用于释放操作系统信号量对象。 |
bool LockSemaphore(
SysSemaphoreObject& semaphore);
|
此函数用于获取对由信号量保护的临界区的访问。 |
bool UnlockSemaphore(
SysSemaphoreObject& semaphore,
int count);
|
此函数用于释放对由信号量保护的临界区的访问。 |
bool MakeEvent(
SysEventObject& event,
bool intialState);
|
此函数用于创建事件的操作系统对象并进行初始化。 |
bool DeleteEvent(
SysEventObject& event);
|
此函数用于释放操作系统事件对象。 |
bool WaitForEvent(
SysEventObject& event);
|
此函数用于阻塞调用线程,直到事件达到已触发状态。当调用线程被释放时,事件会重新设置为非触发状态。 |
bool SignalEvent(
SysEventObject& event);
|
此函数用于将事件设置为已触发状态。 |
同步宏
ATOMIC_INC(VALUE)
|
原子地将 VALUE 加一,并返回新值。 |
ATOMIC_DEC(VALUE)
|
原子地将 VALUE 减一,并返回新值。 |
SPINLOCK(LOCK)
|
用自旋锁保护临界区。LOCK 必须是 int 类型变量。要释放自旋锁,应将 LOCK 变量设置为 0。 |
DEFINE_SYNC_CLASS
|
此宏在类中插入了同步类实例访问所需的成员。LOCK_OBJECT 和 LOCK_THIS_OBJECT 宏用于同步对对象的访问。 |
LOCK(LOCK_NAME)
|
当线程退出 LOCK_OBJECT 指定的作用域时,此宏用于获取对由同步对象(GaSectionLock 或 GaCriticalSection)保护的临界区的访问。解锁对对象的访问可以通过调用 UNLOCK(LOCK_NAME) 宏来实现。 |
UNLOCK(LOCK_NAME)
|
当线程退出 LOCK_OBJECT 指定的作用域时,此宏用于释放对同步对象(GaSectionLock 或 GaCriticalSection)的访问。 |
LOCK_OBJECT(LOCK_NAME,OBJECT)
|
此宏通过实例化一个名为 LOCK_NAME 的 GaSectionLock 对象来获取对对象的访问权限,并阻止并发访问。当执行离开指定 LOCK_OBJECT 的作用域时,GaSectionLock 对象将被销毁,对锁定对象的访问将被释放。在离开作用域之前解锁对对象的访问可以通过调用 UNLOCK(LOCK_NAME) 宏来完成。 |
LOCK_THIS_OBJECT(LOCK_NAME)
|
此宏通过声明并实例化一个名为 LOCK_NAME 的 GaSectionLock 对象来获取对当前对象的访问权限,并阻止并发访问。当执行离开指定 LOCK_OBJECT 的作用域时,GaSectionLock 对象将被销毁,对当前对象的访问将被释放。在离开作用域之前解锁对对象的访问可以通过调用 UNLOCK(LOCK_NAME) 宏来完成。 |
要使用 LOCK_OBJECT 和 LOCK_THIS_OBJECT 宏来同步对对象的访问,该对象所属的类必须使用 DEFINE_SYNC_CLASS 宏,该宏注入这些宏所需的成员。
注入的成员是
mutable GaCriticalSection _synchronizator属性和GaCriticalSection* GACALL GetSynchronizator() const方法
class SomeClass
{
DEFINE_SYNC_CLASS
// rest of the class declaration
};
用户可以使用宏来同步对类实例的访问。
void SomeMethod()
{
SomeClass obj;
// Declares GaSectionLock object obj_lock that
// use critical section object embedded into obj object
LOCK_OBJECT( obj_lock, obj );
// ...critical section code...
// lock is released automatically
// by destroying obj_lock object
}
void SomeClass::SomeMethod()
{
// Declares GaSectionLock object obj_lock that
// use critical section object embedded into this object
LOCK_THIS_OBJECT( obj_lock );
// ...critical section code...
// lock is released automatically
// by destroying obj_lock object
}
如果临界区在作用域结束之前结束,则可以使用 UNLOCK 宏来解锁临界区对象。
void SomeClass::SomeMethod()
{
// Declares GaSectionLock object obj_lock that
// use critical section object embedded into this object
LOCK_THIS_OBJECT( obj_lock );
// ...critical section code...
// release lock on critical section object
UNLOCK( obj_lock );
// ...non-critical code...
// section can be locked again using same lock object
LOCK( obj_lock );
// ...critical section...
}
可以通过直接在临界区对象上使用 LOCK 和 UNLOCK 宏来锁定临界区,而无需使用 GaSectionLock 对象。
// ...
GaCriticalSection _criticalSection;
// ...
void SomeMethod()
{
// lock critical section object
LOCK( _criticalSection );
// ...critical section code...
// release lock
UNLOCK( _criticalSection );
}
目录
目录用于存储可用的遗传操作。每种类型的遗传操作都有自己的目录。遗传操作以对[操作名称和指向操作对象的指针]的形式存储在目录中。操作名称在目录中必须是唯一的。用户可以通过指定操作名称来获取指向操作对象的指针[仿函数]。GaCatalogue 模板类管理目录。

GaCatalogue 类GaCatalogueEntry 类用于存储指向遗传操作对象的指针以及它在目录中注册的名称。
Register 和 Unregister 方法将遗传操作添加到目录或从中移除。GetEntryData 方法按名称查找遗传操作。
遗传算法库定义了八个内置目录
GaCrossoverCatalogueGaMutationCatalogueGaFitnessComparatorCatalogueGaSelectionCatalogueGaCouplingCatalogueGaReplacementCatalogueGaScalingCatalogueGaStopCriteriaCatalogue
在使用特定类型操作的目录之前,必须调用 MakeInstance 方法。目录不再需要后应调用 FreeInstance。对于内置目录,这些方法在库的初始化和最终化期间被调用。对于用户定义的目录,用户必须手动调用这些方法。
// using built-in catalogue
GaSelectionOperation* select =
GaSelectionCatalgoue::Instance().GetEntryData(
"GaRandomSelect" );
// user-defined catalogue
GaCatalgoue<UserOperationType>::MakeInstance();
// register
GaCatalgoue<UserOperationType>::Instance().Register(
"UserOperation1", new UserOperation1() );
GaCatalgoue<UserOperationType>::Instance().Register(
"UserOperation2", new UserOperation2() );
// retrieve data
UserOperationType* operation =
GaCatalgoue<UserOperationType>::Instance().GetEntryData(
"UserOperation1" );
// unregister
GaCatalgoue<UserOperationType>::Instance().Unregister(
"UserOperation1");
// free resources used by catalogue
GaCatalgoue<UserOperationType>::FreeIntance();
目录管理已注册对象使用的内存。
随机数生成器
GaRandomGenerator 类实现了 RANROT 算法来生成随机数。它一次生成一个 64 位宽的整数[两个 32 位整数]。库中的所有其他内置随机数生成器都建立在此类的基础上。
| 数据类型 | Interval | 类名 | 全局对象 |
int |
0-2147483647 | GaRandomInteger |
GaGlobalRandomIntegerGenerator |
float |
(0, 1) | GaRandomFloat |
GaGlobalRandomFloatGenerator |
double |
(0, 1) | GaRandomDouble |
GaGlobalRandomDoubleGenerator |
bool |
true 或 false | GaRandomBool |
GaGlobalRandomBoolGenerator |

这些随机数生成器类具有在预定义区间内、在预定义最小值和用户定义最大值之间以及在用户定义最小值和最大值之间生成数字的方法。GaRandomBool 具有两个额外的方法,用于指定生成 true 值的概率。
染色体表示
遗传算法库定义了几个接口,允许染色体与内置的交叉和变异操作一起使用。
GaMutableCode 接口应由支持对其代码中的值进行随机翻转或反转的染色体实现。此接口定义了两个方法:Invert 和 Flip。Invert 方法应选择定义数量的染色体代码值并反转它们。Flip 方法应更改随机定义的染色体代码值的数量。

GaMutableCode 接口GaSwapableCode 接口应由能够交换染色体代码值块的染色体实现。此接口定义了 Swap 方法。
![]()
GaSwapableCode 接口GaSizableCode 接口应由能够更改其代码中值数量的染色体实现。Insert 方法应在指定位置插入定义数量的随机值到染色体的代码中。Remove 方法应从染色体的代码中指定位置移除一个值块。

GaSizableCode 接口GaMultiValueCode 接口应由其代码中可以具有多个值的染色体实现。此接口定义了将值从染色体的代码提取到缓冲区以及从值缓冲区初始化染色体的方法。MakeBuffer 方法应创建一个可以存储指定数量值的缓冲区对象并返回指向该对象的指针。FillBuffer 应将指定位置的值块复制到缓冲区。FromBuffer 方法应使用提供的缓冲区中的值初始化染色体的代码。

GaMultiValueCode 接口GaCodeValuesBuffer 类用于管理用于存储染色体代码值的缓冲区。缓冲区可以用于存储来自多个染色体的值,因此适合在交叉操作中组合染色体。

GaCodeValuesBuffer 类GaArithmeticalCode 接口应由支持对其代码值进行算术运算的染色体实现。此接口定义了基本算术运算符[+、-、*、/]以及一个用于查找中点的函数。

GaArithmeticalCode 接口GaCodeValue 接口应由包装染色体代码值数据类型的类实现。此接口定义了 FormBuffer 方法,该方法应从提供的缓冲区中提取单个值[在指定位置]。Initialize 方法在实现时,应随机初始化一个包装值。

GaCodeValue 接口在大多数情况下,染色体代码中的值具有约束[域]。这些约束在库中通过值集来实现。GaDomainChromosome 类使用一个 CCB,该 CCB 存储一个指向值集的指针,该值集定义了染色体代码中值的约束。

GaDomainChromosome 和 GaChromosomeDomainBlock 类GaValuSet 是库中值集的基础类。

GaValueSet 类GenerateRandom 方法随机选择一个值集中的值并返回它。Inverse 方法返回一组反转值中的对应值。Belongs 方法如果指定值属于值集,则返回 true。ClosestValue 返回最接近指定值并且属于值集的值。
每个值集都有一个值组[称为原始值]和一个对应的反转值组[称为反转值]。_viceVersa 成员定义了对反转值的处理。如果设置为 true,则反转值被视为值集的有效成员,这意味着反转值可以与值集中定义的所有操作以与原始值相同的方式使用。
GaSingleValueSet 模板类表示只有一个原始值和一个反转值的值集。此值集适用于可以具有两种可能状态的染色体代码的值。

GaSingleValueSet 类GaMultiValueSet 模板类表示具有多个值的值集。原始值组中的每个值都有一个精确对应的反转值。不允许原始值重复。只有当 _viceVersa 设置为 false 时,反转值才能重复。

GaMultiValueSet 类GaIntervalValueSet 模板类表示一个值集,其中包含指定区间内的连续值。区间边界由 GaValueIntervalBounds 模板类定义。集合中值的类型必须定义 +、-、>、>=、< 和 <= 运算符。用户必须提供实现 GaRandom 接口的随机值生成器。

GaIntervalValueSet 类GaUnboundValueSet 模板类应在染色体代码的值没有附加约束时使用。存储值的类型必须定义一元 - 运算符。用户必须提供实现 GaRandom 接口的随机值生成器。此值集生成的值的范围仅由提供的随机生成器确定。

GaUnboundValueSet 类GaCombinedValueSet 可用于将两个或多个值集合并为一个集。

GaCombinedValueSet 类GaSingleValueChromosome 模板类可用于表示单个值解决方案的染色体。它可以是单个实数或整数,或任何其他类型的值。此类仅实现 GaMutableCode 接口。由于 SingleValueChromosome 继承自 GaDomainChromosome 类,因此可以使用值集来控制值的域。
GaSVAChromosome 模板类适用于支持算术运算的染色体,因为它实现了 GaArithmeticalCode。染色体值的类型必须定义 +、-、*、/ 运算符以及与整数类型右侧操作数的 / 运算符。这允许染色体与内置的算术交叉操作一起使用。

GaSingleValueChromosome 类GaMultiValueValueChromosome 模板类可用于需要多个值来表示解决方案的染色体。此类实现了 GaMutableCode、GaSwapableCode、GaSizableCode 和 GaMultiValueCode 接口。GaChromosomeValue 类用于将值注入染色体代码或从中提取值。
GaMVAChromosome 模板类在 GaSingleValueValueChromosome 的基础上扩展了 GaMultiValueValueChromosome,正如 GaSVAChromosome 扩展 GaSingleValueValueChromosome 一样。
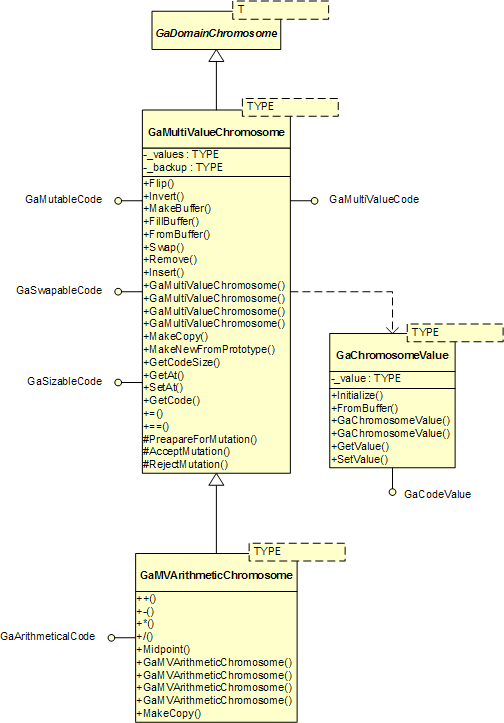
GaMultiValueChromosome 类GaBinaryChromosome 模板类实现了使用二进制编码表示解决方案的染色体。此类具有一组方法,可将内置类型编码为二进制流,并将二进制流解码为原始值。GaBinaryChromosome 使用 GaBinaryChromosomeParams 类作为染色体参数。此类定义了创建染色体时设置概率的状态。
GaBit 类用于将位注入染色体代码或从中提取位。

GaBinaryChromosome 类这些类位于 Chromosome::Representation 命名空间中。
内置适应度比较器
内置适应度比较器位于 Chromosome::FitnessComparators 命名空间中。

有两个适应度比较器
GaMaxFitnessComparator- 用于最大化适应度值的遗传算法,GaMinFitnessComparator- 用于最小化适应度值的遗传算法。
内置交叉操作
内置交叉操作位于 Chromosome::Crossover 命名空间中。

GaMutiValueCrossover 类实现了一个交叉操作,该操作通过选择 N 个交叉点来创建子代,然后依次复制父代的值,每次到达交叉点时切换源父代。此操作需要实现 GaMultiValueCode 接口的染色体。

GaMutiValueCrossover 操作的结果GaMidpointCrossover 类实现了一个交叉操作,该操作通过调用选定父代的 Midpoint 方法来创建子代。此操作需要实现 GaArithmeticalCode 接口的染色体。

GaMidpointCrossover 操作的结果[多值染色体,值为 int 类型]GaAddCrossover 调用父代染色体的 operator+,GaSubCrossover 调用 operator-。

GaAddCrossover 操作的结果[多值染色体,值为 int 类型]
GaSubCrossover 操作的结果[多值染色体,值为 int 类型]内置变异操作
内置变异操作位于 Chromosome::Mutation 命名空间中。

GaSwapMutation 类实现了一个变异操作,该操作选择染色体代码中的两个值块并交换它们在代码中的位置[交换的最大值数量由染色体参数中指定的变异大小定义]。

GaSwapMutation 操作的结果GaInvertMutation 类实现了一个变异操作,该操作选择 N 个值[最大值数量由染色体参数中指定的变异大小定义]并使用染色体定义的值集的 Invert 方法反转它们的值。此操作需要实现 GaMutableCode 接口的染色体。

GaInvertMutation 操作的结果GaFlipMutation 类实现了一个变异操作,该操作选择 N 个值[最大值数量由染色体参数中指定的变异大小定义]并使用染色体定义的值集的 GenerateRandom 方法随机设置它们的值。此操作需要实现 GaMutableCode 接口的染色体。

GaFlipMutation 操作的结果内置选择操作
内置选择操作位于 Population::SelectionOperations 命名空间中。

GaSelectBest 和 GaSelectWorst 选择操作GaSelectBest 类实现了一个选择操作,该操作选择种群中最好的 N 个染色体[由操作参数中定义的选择大小]。如果种群未排序,则该操作只能选择种群中最佳染色体排序组中的那些染色体。

GaSelectRouletteWheel 和 GaSelectTournament 操作GaSelectRouletteWheel 类实现了一个基于适应度值选择染色体的选择操作。染色体的适应度值用于计算它们的选择概率。当遗传算法最大化适应度时,更高的适应度意味着更高的选择概率。当遗传算法最小化适应度时,较低的适应度意味着更高的选择概率。该操作需要一个已排序的种群。如果种群定义了缩放操作,则选择使用缩放后的适应度值;否则,它使用原始适应度值。此选择操作可以选择单个父代多次,这可能对某些替换操作造成问题。为避免此问题,GaSelectRouletteWheel 使用 GaSelectDuplicatesParams 类作为其参数来控制选择结果集中的重复项。
GaSelectTournament 使用与 GaSelectRouletteWheel 类似的方法。它为选择结果集中的单个位置执行 N 次[由操作参数定义]“轮盘赌”选择。选择的染色体中最好的一个被放入结果集。重复此过程以选择所有父代。该操作使用 GaSelectTournamentParam 类作为其参数。

GaSelectRandom 和 GaSelectRandomBest 操作GaSelectRandom 类实现了一个随机选择父代的选择操作。该操作可以选择单个父代多次,这可能对某些替换操作造成问题。为避免此问题,GaSelectRandom 使用 GaSelectDuplicatesParams 类作为其参数来控制选择结果集中的重复项。
GaSelectRandomBest 的工作方式与 GaSelectRandom 相同,但它选择比参数定义的父代更多的父代;然后,它截断结果集以使其适合所需的选择大小,只留下最佳父代。GaRandomBestParams 类由该操作使用,它定义了截断前要选择的父代数量。
内置耦合操作
内置耦合操作位于 Population::CouplingOperations 命名空间中。
![Built-in Coupling Operations [1]](https://cloudfront.codeproject.com/recipes/geneticlibrary/gacouplingoperation1.png)
GaSimpleCoupling 操作从选择结果集中取前两个父代,然后使用交叉操作产生两个后代染色体,每个父代绑定一个子代,然后它取接下来的两个父代,依此类推……如果所有父代都已使用,但仍需产生更多子代,则操作会从选择结果集开头重新开始,直到产生足够多的子代。此耦合操作使用 GaCouplingParams 类作为其参数。

GaSimpleCoupling 操作![Built-in Coupling Operations [2]](https://cloudfront.codeproject.com/recipes/geneticlibrary/gacouplingoperation2.png)
GaCrossCoupling 操作顺序地从选择结果集中获取父代。如果所有父代都已使用,但仍需产生更多子代,则操作会从开头重新开始,直到产生足够多的子代。

GaCrossCoupling 操作GaInverseCoupling 操作顺序地从选择结果中取第一个父代,第二个父代是与最后一个染色体在选择结果集中的距离等于第一个父代与结果集第一个染色体距离的染色体。如果所有父代都已使用,但仍需产生更多子代,则操作会从开头重新开始,直到产生足够多的子代。

GaInverseCoupling 操作GaRandomCoupling 操作顺序地从选择结果集中取第一个父代,然后随机选择第二个父代。如果所有父代都已作为第一个父代使用,但仍需产生更多子代,则操作会从开头重新开始,直到产生足够多的子代。

GaRandomCoupling 操作GaBestAlwaysCoupling 操作始终选择选择结果集中适应度值最高的染色体作为第一个父代,然后顺序选择第二个父代。如果所有父代都已使用,但仍需产生更多子代,则操作会从开头重新开始,直到产生足够多的子代。

GaBestAlwaysCoupling 操作当选择两个父代时,这些操作使用交叉操作产生指定数量的子代。然后,它们选择在产生的子代中适应度值最高的子代,将子代存储在耦合结果集中,并将父代绑定到子代。这些耦合操作使用 GaMultipleCrossoverCouplingParams 类作为参数来控制每对父代产生的子代数量。
内置替换操作
内置替换操作位于 Population::ReplacementOperations 命名空间中。

GaReplaceWorst 和 GaReplacBest 操作GaReplaceWorst 操作用来自提供的耦合结果集的后代染色体替换种群中最差的染色体。如果种群未排序,则该操作只能替换种群中最差排序组中存储的染色体。此操作使用 GaReplacementParams 类作为其参数。
GaReplaceBest 操作的工作方式与 GaReplaceWorst 相同,但它替换种群中最好的染色体。

GaReplaceParents 和 GaReplaceRandom 操作GaReplaceParents 操作替换耦合结果集中后代染色体的父代。如前所述,耦合操作将染色体父代的信息存储在耦合结果集中。如果使用此操作,则选择操作不应选择重复的染色体,并且耦合操作不应将一个父代绑定到多个子代。
GaReplaceRandom 操作随机选择将被替换的染色体。
这两个操作可能会从种群中移除最好的染色体;为防止这种情况,它们实现了精英主义机制。GaReplaceElitismParams 类被这些操作使用,并定义了种群中不被替换操作影响的最佳染色体数量。
内置缩放操作
内置缩放操作位于 Population::ScalingOperations 命名空间中。

GaWindowScaling 和 GaNormalizationScaling 操作GaWindowScaling 操作通过从最差染色体的适应度中减去被缩放染色体的适应度来计算缩放后的适应度[scaled_fitness = chromosome_fitness - worst_chromosome_fitness]。
GaNoramlizationScaling 操作根据染色体的排名计算缩放后的适应度[scaled_fitness = population_size - chromosome_rank]。它需要一个已排序的种群。
这些操作不需要参数。

GaLinearScaling 和 GaExponentialScaling 操作GaLinearScaling 操作使用线性变换缩放适应度值:scaled_fitness = a * fitness + b [a 和 b 是缩放系数]。a 和 b 的计算方法如下
if( min_f > ( factor * avg_f - max_f ) / factor - 1 )
{
a = avg_f / ( avg_f - min_f );
b = -1 * min_f * avg_f / ( avg_f - min_f );
}
else
{
a = ( factor - 1 ) * avg_f / ( max_f - avg_f );
b = avg_f * ( max_f - factor * avg_f ) / ( max_f - avg_f );
}
min_f- 种群中最差染色体的适应度值max_f- 种群中最佳染色体的适应度值avg_f- 种群中所有染色体的平均适应度值factor- 缩放因子 [用于乘以平均适应度,从而确定种群中最佳染色体的缩放适应度值]
此操作不能处理负适应度值。
GaExponentialScaling 操作通过将染色体的适应度值提高到指定的幂 [由种群参数指定] 来计算缩放适应度。
这两个操作都使用 GaScaleFactorParams 类作为其参数。
内置停止条件操作
内置停止条件操作位于 Population::StopCriterias 命名空间。

GaGenerationCriteria 和 GaGenerationCriteriaParams 类GaGenerationCriteria 用于在遗传算法达到所需的代数时停止。它使用 GaGenerationCriteriaParams 类作为参数。

GaFitnessCriteria 和 GaFitnessCriteriaParams 类GaFitnessCriteria 根据算法的适应度值统计信息 [原始或缩放的,例如最佳染色体的适应度、平均适应度或最差染色体的适应度] 来决定何时停止算法。GaFitnessCriteriaComparison 枚举定义了可用于将所需的适应度值与指定限制进行比较的比较类型。GaFitnessCriteria 使用 GaFitnessCriteriaParams 类作为其参数。

GaFitnessProgressCriteria 和 GaFitnessProgressCriteriaParams 类GaFitnessProgressCriteria 根据算法适应度值统计信息的进展来决定何时停止算法。此标准衡量并跟踪算法代数中所需值的进展。如果在定义的代数数量之后,算法在一代中未能取得所需的进展,则该标准指示算法停止。GaFitnessProgressCriteriaParams 类代表此标准的参数。参数类还存储算法进展的历史记录。
内置遗传算法
内置遗传算法位于 Population::SimpleAlgorithms 命名空间。

GaSimplemAlgorithm 类实现了一个具有不重叠种群的遗传算法。用户在创建遗传算法时需要提供一个种群对象 [不一定需要初始化]。该算法实现了精英主义,并且保存的染色体数量由算法的参数 [GaSimpleAlgorithmParams 类] 定义。算法的工作方式如下:
- 创建提供的种群对象的副本
- 初始化提供的种群对象
- 选择父代并产生子代染色体
- 从当前种群复制最佳染色体 [精英主义]
- 将子代染色体插入新种群
- 检查停止条件 [达到则退出]
- 切换种群对象并返回步骤 3。
GaIncrementalAlgorithm 类实现了一个使用重叠种群的遗传算法,并且每代只替换少量染色体 [使用替换操作]。用户在创建遗传算法时需要提供一个种群对象 [不一定需要初始化]。算法的工作方式如下:
- 初始化提供的种群对象
- 选择父代并产生子代染色体
- 用子代替换旧染色体
- 检查停止条件 [达到则退出]
- 切换种群对象并返回步骤 2。
示例
用户必须执行以下步骤来使用此库构建遗传算法
- 选择染色体的表示
- 定义适应度操作
- 选择交叉和变异操作以及适应度比较器
- 选择选择、耦合、替换和缩放操作
- 选择算法类型和停止条件
示例 1:寻找 f(x, y) = 5*x*sin(x) + 1.1*y*sin(y); x, y 在区间 (0, 10) 的最小值
重要提示:在使用 GAL 之前,必须调用 GaInitialize。此外,在退出应用程序之前,必须调用 GaFinalize。
最简单的方法是选择支持算术运算的多值染色体表示 [Chromosome::Representation::GaMVArithmeticChromosome<double> 类]。
选择染色体表示后,用户必须定义适应度操作。
class fFitness : public GaFitnessOperation
{
public:
virtual float GACALL operator() (const GaChromosome*
chromosome) const
{
const vector<double>& vals=
dynamic_cast<const GaMVArithmeticChromosome<double>*>
(chromosome)->GetCode();
return 5*vals[0]*sin(vals[0])+1.1*vals[1]*sin(vals[1]);
}
virtual GaParameters* GACALL MakeParameters() const { return NULL; }
virtual bool GACALL CheckParameters(const GaParameters&
parameters) const { return true; }
};
fFitness 类继承自 GaFitnessOperation 类,并重写了 operator(),该函数通过评估实际的数学函数来计算染色体的适应度值。
下一步是构建一个染色体配置块 (CCB),其中包含
- 染色体参数的指针
- 遗传操作函数对象 [交叉、变异、适应度操作和适应度比较器] 的指针
- 定义 *
x* 和 *y* 变量域的值集的指针
Chromosome::Representation::GaValueInterval<T> 类用作染色体的值集,因为 *x* 和 *y* 变量的域是连续区间 (0, 10)。GaIntervalValueSet 需要四个边界 [低和高边界用于指定原始值的区间,低和高边界用于指定反转值的区间] 和一个随机值生成器。
GaValueIntervalBound<double /> valueInt(0,10);
GaValueIntervalBound<double /> invValueInt(0,10);
GaValueInterval<double /> valueSet(valueInt,invValueInt,
GaGlobalRandomDoubleGenerator,false);
CCB 应该为
fFitness fitnessOperation;
GaChromosomeDomainBlock<double> configBlock(&valueSet,
GaCrossoverCatalogue::Instance().GetEntryData(
"GaMultiValueCrossover"),
GaMutationCatalogue::Instance().GetEntryData("GaFlipMutation"),
&fitnessOperation,
GaFitnessComparatorCatalogue::Instance().GetEntryData(
"GaMinFitnessComparator"),
&chromosomeParams);
CCB 定义为使用 GaMultiValuesCrossover 和 GaFlipMutation 操作。指定 GaMinFitnessComparator 是因为算法的目的是寻找函数的最小值。
定义 CCB 后,用户可以构建原型染色体
GaMVArithmeticChromosome<double> prototype(2,&configBlock);
除了原型染色体之外,用户还必须在创建种群对象之前定义种群参数
- 种群大小:30
- 可调整大小的种群:否 [使用了增量算法,该算法不需要可调整大小的种群]
- 种群已排序:是
- 使用缩放的适应度进行排序:否
- 最佳染色体跟踪:0 [种群已排序]
- 最差染色体跟踪:0 [种群已排序]
GaPopulationParameters populationParams(30,false,true,false,0,0);
此种群对象使用默认配置,但更改了排序比较器
- 选择操作:
GaSelectRouletteWheel - 选定的染色体数量:2
- 耦合操作:
GaInverseCoupling - 产生的子代:2
- 替换操作:
GaReplaceWorst - 替换的染色体:2
- 缩放操作:无
- 排序比较器:
GaMaxFitnessComparator[默认] 更改为GaMinFitnessComparator
现在一切都准备好创建种群对象了
GaPopulationConfiguration populationConfig;
populationConfig.SetParameters(populationParams);
populationConfig.SetSortComparator(
&configBlock.GetFitnessComparator());
GaPopulation population( &prototype, &populationConfig );
此示例使用增量遗传算法 [GaIncrementalAlgorithm 类]。要创建算法对象
GaMultithreadingAlgorithmParams algorithmParams(1);
GaIncrementalAlgorithm algorithm(&population,algorithmParams);
其中用户指定遗传算法将操作的种群以及算法的参数。算法参数的构造函数接受工作线程的数量。
当用户为这类问题构建遗传算法时,无法确切知道算法的终止条件。在这种情况下,使用基于进化过程持续时间或其进展的停止条件很方便。其中一种停止条件是基于代数的停止条件。该示例仅使用一个线程,因为算法每代只产生少量新的染色体。
GaGenerationCriteriaParams criteriaParams(100000);
algorithm.SetStopCriteria(
GaStopCriteriaCatalogue::Instance().GetEntryData(
"GaGenerationCriteria"),&criteriaParams);
停止条件参数的构造函数接受算法停止之前的代数数量。
为了监控进化过程,用户必须向遗传算法指定一个观察者对象。
class fObserver : public GaObserverAdapter
{
virtual void GACALL NewBestChromosome(const GaChromosome&
newChromosome,const GaAlgorithm& algorithm)
{
const vector<double>& vals=
dynamic_cast<const GaMVArithmeticChromosome<double>&>
(newChromosome).GetCode();
cout << "New chromosome found:\n";
cout << "Fitness: " << newChromosome.GetFitness() << endl;
cout << "x: " << vals[0] << " y: " << vals[1] << endl;
}
virtual void GACALL EvolutionStateChanged(GaAlgorithmState
newState,const GaAlgorithm& algorithm)
{
if(newState==GAS_RUNNING)
cout << "start\n";
else if(newState==GAS_CRITERIA_STOPPED)
cout << "end";
}
};
注册观察者
fObserver observer;
algorithm.SubscribeObserver(&observer);
启动算法
algorithm.StartSolving(false);
StartSolving 的参数定义了算法是继续先前暂停的进化过程 [true] 还是开始一个全新的过程 [false]。
示例 2: 模式匹配
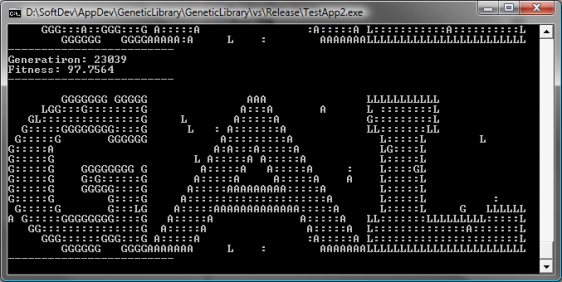
此示例实现了一个试图猜测字符序列的遗传算法。该示例定义了这个字符序列
const char pattern[] =
" GGGGGGGGGGGGG AAA LLLLLLLLLLL "
" GGG::::::::::::G A:::A L:::::::::L "
" GG:::::::::::::::G A:::::A L:::::::::L "
" G:::::GGGGGGGG::::G A:::::::A LL:::::::LL "
" G:::::G GGGGGG A:::::::::A L:::::L "
"G:::::G A:::::A:::::A L:::::L "
"G:::::G A:::::A A:::::A L:::::L "
"G:::::G GGGGGGGGGG A:::::A A:::::A L:::::L "
"G:::::G G::::::::G A:::::A A:::::A L:::::L "
"G:::::G GGGGG::::G A:::::AAAAAAAAA:::::A L:::::L "
"G:::::G G::::G A:::::::::::::::::::::A L:::::L "
" G:::::G G::::G A:::::AAAAAAAAAAAAA:::::A L:::::L LLLLLL"
" G:::::GGGGGGGG::::G A:::::A A:::::A LL:::::::LLLLLLLLL:::::L"
" GG:::::::::::::::G A:::::A A:::::A L::::::::::::::::::::::L"
" GGG::::::GGG:::G A:::::A A:::::A L::::::::::::::::::::::L"
" GGGGGG GGGGAAAAAAA AAAAAAALLLLLLLLLLLLLLLLLLLLLLLL";
const int patternSize=sizeof(pattern)-1;
使用的符号是:*G*、*A*、*L*、*:* 和一个空格。
遗传算法使用 Chromosome::Representation::GaMVArithmeticChromosome<double> 染色体表示,其值域由 Chromosome::Representation::GaMultiValueSet<char> 类定义。
GaMultiValueSet<char> valueSet(false);
valueSet.Add("GAL: "," ",5);
适应度操作计算匹配字符的百分比,并将该数字作为染色体的适应度值返回
class pFitness : public GaFitnessOperation
{
public:
virtual float GACALL operator()(const GaChromosome*
chromosome) const
{
const vector<char>& v=
dynamic_cast<const GaMultiValueChromosome<char>*>
(chromosome)->GetCode();
int score=0;
for(int i=0;i<patternSize;i++)
{
if(v[i]==pattern[i])
score++;
}
return (float)score/patternSize*100;
}
virtual GaParameters* GACALL MakeParameters() const
{ return NULL; }
virtual bool GACALL CheckParameters(const GaParameters&
parameters) const { return true; }
};
CCB 的样子与前一个示例中的 CCB 类似,只是它使用了一个新的适应度操作和一个不同的适应度比较器,因为现在的目标是最大化适应度
pFitness fitnessOperation;
GaChromosomeDomainBlock<char /> configBlock(&valueSet,
GaCrossoverCatalogue::Instance().GetEntryData(
"GaMultiValueCrossover"),
GaMutationCatalogue::Instance().GetEntryData("GaFlipMutation"),
&fitnessOperation,
GaFitnessComparatorCatalogue::Instance().GetEntryData(
"GaMaxFitnessComparator"),
&chromosomeParams);
原型染色体
GaMultiValueChromosome prototype( patternSize, &configBlock );
此示例使用具有不重叠种群的遗传算法,并且它会产生整个种群。为了增加产生的染色体的多样性,增加了选定染色体的数量。请注意,这种类型的算法需要一个可调整大小的种群。种群对象及其配置
GaPopulationParameters populationParams(30,true,true,false,0,0);
GaPopulationConfiguration populationConfig;
populationConfig.SetParameters(populationParams);
populationConfig.SetSortComparator(
&configBlock.GetFitnessComparator());
populationConfig.Selection().GetParameters().SetSelectionSize(6);
GaPopulation population(&prototype, &populationConfig);
如前所述,此示例使用 Algorithm::SimpleAlgorithms::GaSimpleAlgorithm 类作为遗传算法。
GaSimpleAlgorithmParams algorithmParams(10,2);
GaSimpleAlgorithm algorithm(&population,algorithmParams);
参数构造函数的第一参数是精英主义深度,第二参数是工作线程的数量。该算法每代产生的染色体比前一个多得多,因此适合并行化。
在此示例中,确切的终止条件是已知的:当算法找到适应度值为 100 [100% 匹配] 的染色体时。正确的停止条件是 Algorithm::StopCriterias::GaFitnessCriteria
GaFitnessCriteriaParams criteriaParams(100,GFC_EQUALS_TO,
GSV_BEST_FITNESS);
algorithm.SetStopCriteria(
GaStopCriteriaCatalogue::Instance().GetEntryData(
"GaFitnessCriteria"),
&criteriaParams);
算法的观察者会显示找到的最佳染色体。
class pObserver : public GaObserverAdapter
{
public:
virtual void GACALL NewBestChromosome(const GaChromosome&
newChromosome,const GaAlgorithm& algorithm)
{
const vector<char>& v=
dynamic_cast<const GaMultiValueChromosome<char>&>
(newChromosome).GetCode();
cout<<"Generatiron: "<<
algorithm.GetAlgorithmStatistics().GetCurrentGeneration()
<<endl;
cout<<"Fitness: "<<newChromosome.GetFitness();
cout<<"\n-------------------------\n";
for(int i=0;i<v.size();i++)
{
if(!(i%78))
cout<<endl;
cout<<v[i];
}
cout<<"\n-------------------------\n";
}
virtual void GACALL EvolutionStateChanged(GaAlgorithmState
newState,const GaAlgorithm& algorithm)
{
if(newState==GAS_CRITERIA_STOPPED)
cout<<"end.";
}
};
观察者的订阅与前一个示例相同。
pObserver observer;
algorithm.SubscribeObserver( &observer );
进化开始
algorithm.StartSolving(false);
示例 3: 旅行商问题

染色体是按访问顺序排列的城市 [TspCity 类的对象指针] 数组。它由 TspChromosome 类实现。该类继承自 GaMultiValueChromosome,通过重写 MakeFromPrototype 方法来实现自定义染色体初始化。此方法将城市复制到染色体的代码中,然后对其位置进行混洗。该类还重写了 MakeCopy 方法并定义了复制构造函数。
class TspChromosome : public GaMultiValueChromosome<const TspCity*>
{
public:
TspChromosome(GaChromosomeDomainBlock<const TspCity*>* configBlock) :
GaMultiValueChromosome(configBlock) { }
TspChromosome(const TspChromosome& chromosome,
bool setupOnly) :
GaMultiValueChromosome<const TspCity*>(chromosome, setupOnly) { }
virtual GaChromosomePtr GACALL MakeCopy(bool setupOnly) const
{ return new TspChromosome( *this, setupOnly ); }
virtual GaChromosomePtr GACALL MakeNewFromPrototype() const;
int GACALL GetCityPosition(const TspCity* city) const;
};
使用简单的单点或多点交叉操作将生成大量无效解,从而降低算法的性能和结果。为防止生成无效解,算法使用自定义交叉操作。该操作从一个父代中选取一个随机城市,并将其复制到子代染色体。然后,它查找与所选城市连接的城市 [在两个父代中],并取最近的一个 [并将其复制到子代染色体],如果尚未被采用。如果操作选择另一个连接的城市,则该城市已被采用。如果所有连接的城市都已被采用,则该操作会随机选择一个尚未被采用的城市。然后,交叉操作使用该城市以相同方式扩展路径。重复该过程以选择所有城市。TspCrossover 类实现了此交叉操作。
class TspCrossover : public GaCrossoverOperation
{
public:
virtual GaChromosomePtr GACALL operator ()(
const GaChromosome* parent1,
const GaChromosome* parent2) const;
virtual GaParameters* GACALL MakeParameters() const { return NULL; }
virtual bool GACALL CheckParameters(
const GaParameters& parameters) const { return true; }
private:
inline void SelectNextCity(const TspCity* previousCity,
const TspCity** currentBestNextCity,
const TspCity* nextCity) const;
};
算法使用内置的 GaSwapMutation 操作。适应度值等于路径长度。TspFitnessOperation 类实现了适应度操作。
class TspFitness : public GaFitnessOperation
{
public:
virtual float GACALL operator ()(
const GaChromosome* chromosome) const;
virtual GaParameters* GACALL MakeParameters() const { return NULL; }
virtual bool GACALL CheckParameters(
const GaParameters& parameters) const { return true; }
};
染色体参数
- 变异概率:3%
- 变异大小:2
- 仅改进变异:否
- 交叉概率:80%
- 交叉点数量:1 [忽略]
CCB
TspCrossoverTspSwapMutationTspFitnessOperationTspMinFitnessComparator- 未定义值集
种群参数
- 种群大小:100
- 可调整大小的种群:否 [使用了增量算法,该算法不需要可调整大小的种群]
- 种群已排序:是
- 使用缩放的适应度进行排序:否
- 最佳染色体跟踪:0 [种群已排序]
- 最差染色体跟踪:0 [种群已排序]
种群配置
GaSelectRandomBest选择,选择 8 个染色体GaSimpleCoupling,产生 8 个子代染色体GaRandomReplaces,每代替换 8 个染色体,精英主义大小为 10 个染色体- 无缩放操作
算法使用 GaFitnessProgressCriteria,因为确切的终止条件是未知的。该标准将在算法无法在超过 50000 代中的 1 代内改进适应度值时停止算法。该遗传算法是增量的。
TSP 类是算法对象的容器。TspCity 类表示并存储有关城市的信息 [例如其坐标和名称]。它具有 GetDistance 方法,用于计算城市之间的距离。TspCities 管理用户输入的城市集合。
示例 4: 课程表

制作课程表的遗传算法已在此 处 描述。在本文中,演示应用程序使用遗传算法库演示了同一问题的解决方案。
Schedule 类定义了染色体的表示。它继承自 GaMultiValueChromosome。
class Schedule : public GaMultiValueChromosome<list<CourseClass*> >
{
friend class ScheduleCrossover;
friend class ScheduleMutation;
friend class ScheduleFitness;
friend class ScheduleObserver;
private:
CourseClassHashMap _classes;
CourseClassHashMap _backupClasses;
// Flags of class requirements satisfaction
mutable vector<bool> _criteria;
public:
Schedule(GaChromosomeDomainBlock<list<CourseClass*> >* configBlock);
Schedule(const Schedule& c, bool setupOnly);
virtual ~Schedule() { }
virtual GaChromosomePtr GACALL MakeCopy(bool setupOnly) const
{ return new Schedule( *this, setupOnly ); }
virtual GaChromosomePtr GACALL MakeNewFromPrototype() const;
virtual void GACALL PreapareForMutation();
virtual void GACALL AcceptMutation();
virtual void GACALL RejectMutation();
// Returns reference to table of classes
inline const hash_map<courseclass*, />& GetClasses() const
{ return _classes; }
// Returns array of flags of class requirements satisfaction
inline const vector<bool>& GetCriteria() const
{ return _criteria; }
// Return reference to array of time-space slots
inline const vector<list<CourseClass*>>& GetSlots() const
{ return _values; }
};
然后定义交叉、变异和适应度操作
class ScheduleCrossover : public GaCrossoverOperation
{
public:
virtual GaChromosomePtr GACALL operator ()(
const GaChromosome* parent1,
const GaChromosome* parent2) const;
virtual GaParameters* GACALL MakeParameters() const
{ return NULL; }
virtual bool GACALL CheckParameters(
const GaParameters& parameters) const { return true; }
};
class ScheduleMutation : public GaMutationOperation
{
public:
virtual void GACALL operator ()(
GaChromosome* chromosome) const;
virtual GaParameters* GACALL MakeParameters() const
{ return NULL; }
virtual bool GACALL CheckParameters(
const GaParameters& parameters) const { return true; }
};
class ScheduleFitness : public GaFitnessOperation
{
public:
virtual float GACALL operator ()(
const GaChromosome* chromosome) const;
virtual GaParameters* GACALL MakeParameters() const
{ return NULL; }
virtual bool GACALL CheckParameters(
const GaParameters& parameters) const { return true; }
};
有关染色体表示和这些操作的更多信息,请参阅 本文。
ScheduleTest 类是遗传算法对象的容器。
ScheduleTest::ScheduleTest()
{
// initialize GAL internal structures
GaInitialize();
// make chromosome parameters
// crossover probability: 80%
// crossover points: 2
// no "improving only mutations"
// mutation probability: 3%
// number of moved classes per mutation: 2
_chromosomeParams = new GaChromosomeParams(
0.03F, 2, false, 0.8F, 2 );
// make CCB with fallowing setup:
// there are no value set
// with ScheduleCrossover, ScheduleMutation and
// ScheduleFitness genetic operations
// set fitness comparator for maximizing fitness value
// use previously defined chromosome's parameters
_ccb = new GaChromosomeDomainBlock<list<courseclass* /> >(
NULL, &_crossoverOperation, &_mutationOperation,
&_fitnessOperation,
GaFitnessComparatorCatalogue::Instance().GetEntryData(
"GaMaxFitnessComparator" ),
_chromosomeParams );
// make prototype of chromosome
_prototype = new Schedule( _ccb );
// make population parameters
// number of chromosomes in population: 100
// population always has fixed number of chromosomes
// population is not sorted
// non-transformed(non-scaled) fitness values are used
// for sorting and tracking chromosomes
// population tracks 5 best and 5 worst chromosomes
GaPopulationParameters populationParams(
100, false, true, false, 2, 0 );
// make parameters for selection operation
// selection will choose 16 chromosomes
// but only 8 best of them will be stored in selection result set
// there will be no duplicates of chromosomes in result set
GaSelectRandomBestParams selParam( 10, false, 16 );
// make parameters for replacement operation
// replace 8 chromosomes
// but keep 5 best chromosomes in population
GaReplaceElitismParams repParam( 10, 2 );
// make parameters for coupling operation
// coupling operation will produce 8 new chromosomes
// from selected parents
GaCouplingParams coupParam( 10 );
// make population configuration
// use defined population parameters
// use same comparator for sorting as comparator used by chromosomes
// use selection operation which randomly selects chromosomes
// use replacement operation which randomly chooses
// chromosomes from population
// which are going to be replaced, but keeps best chromosomes
// use simple coupling
// disable scaling
_populationConfig = new GaPopulationConfiguration( populationParams,
&_ccb->GetFitnessComparator(),
GaSelectionCatalogue::Instance().GetEntryData(
"GaSelectRandom" ), &selParam,
GaReplacementCatalogue::Instance().GetEntryData(
"GaReplaceRandom" ), &repParam,
GaCouplingCatalogue::Instance().GetEntryData(
"GaSimpleCoupling" ), &coupParam,
NULL, NULL );
// make population
// with previously defined prototype of chromosomes
// and population configuration
_population = new GaPopulation( _prototype, _populationConfig );
// make parameters for genetic algorithms
// algorithm will use two workers
GaMultithreadingAlgorithmParams algorithmParams( 2 );
// make incremental algorithm with previously defined population
// and parameters
_algorithm = new GaIncrementalAlgorithm(
_population, algorithmParams );
// make parameters for stop criteria based on fitness value
// stop when best chromosome reaches fitness value of 1
GaFitnessCriteriaParams criteriaParams(
1, GFC_EQUALS_TO, GSV_BEST_FITNESS );
// sets algorithm's stop criteria (base on fitness value)
// and its parameters
_algorithm->SetStopCriteria(
GaStopCriteriaCatalogue::Instance().GetEntryData(
"GaFitnessCriteria" ), &criteriaParams );
// subscribe observer
_algorithm->SubscribeObserver( &_observer );
}
移植、编译和链接遗传算法库
遗传算法库支持以下编译器和平台
| Microsoft C++ | Intel C++ | GCC G++ | Borland C++ | Sun Studio C++ | |||||||||||||||||||
| Windows |
|
|
|
|
|
||||||||||||||||||
| Linux |
|
|
|
|
|
||||||||||||||||||
| Mac OS X |
|
|
|
|
|
||||||||||||||||||
| *BSD |
|
|
|
|
|
||||||||||||||||||
| Solaris |
|
|
|
|
|
||||||||||||||||||
|
|||||||||||||||||||||||
该库包含一组预处理器指令,这些指令根据检测到的编译器和目标操作系统控制编译过程。
Windows 平台
遗传算法库提供两个版本的 Visual Studio 2005 项目。第一个配置为使用 Microsoft C/C++ 编译器,第二个配置为使用 Intel C++ 编译器。项目位于 /vs 目录中。
要将遗传算法库的功能添加到应用程序中,必须将其与应用程序链接。在 Visual Studio 2005 中有两种方法可以做到这一点
- 方法 1
将遗传算法库项目添加到应用程序的解决方案,然后设置对遗传算法库项目的引用。
- 方法 2
- 将 GeneticAlgorithm.lib 添加到 Project Properties->Linker->Additional Dependencies。
- 设置适当的目录,以便 Visual Studio 可以找到库及其头文件。这可以在本地或全局完成。
- 本地
- 将 GeneticLibrary.lib 添加到
Project Properties->Linker->General->Additional Library Directories
- 将遗传算法库源代码目录 (/source) 添加到预处理器搜索路径
Project Properties->C/C++->General->Additional Include Directories
- 将 GeneticLibrary.lib 添加到
- 全局地将目录添加到适当的位置 (Include files 和 Library files)
Tools->Options->Projects and Solutions->VC++ Directories
- 本地
这两个项目的过程相同。
该库可以编译为静态库或动态库 [DLL]。默认情况下,它编译为 DLL;如果将其编译并用作静态库,则必须定义 GENETICLIBRARY_STATIC。
当库编译为 DLL 时,输出文件为 GeneticLibrary.dll 和 GeneticLibrary.lib;如果编译为静态库,则只有 GeneticLibrary.lib。这些文件位于 /build/%configuration%/%compiler% 目录中,其中 %configuration% 是 debug 或 release,%compiler% 是 Microsoft C/C++ 编译器的 msvc,或者 Intel C++ 编译器的 icc_win。必须将 GeneticLibrary.dll 文件复制到应用程序的可执行文件所在的同一目录。
遗传算法库默认链接到通用运行时库 [CRT] 的动态版本。当库链接到 CRT 的动态版本时,应用程序在没有安装 Microsoft Visual C++ 2005 可再发行组件包的机器上可能无法启动。需要注意的是,使用遗传算法库的应用程序必须链接到与库相同的 CRT 版本。
Linux、Mac OS X、Solaris 和 *BSD 平台
可以通过从控制台调用 make 并使用适当的 Makefile 来编译库。在 Solaris 操作系统上,使用 gmake 来编译 GCC G++ 库,使用 dmake 来编译 Sun Studio C++ 库。对于 *BSD 系统,请使用 GNU make [gmake] 而不是 BSD make [make]。
make -f %compiler%_%platform%_%configuration% all
其中 %compiler% 是
- gcc - 用于 GCC G++ 编译器。
- icc - 用于 Intel C++ 编译器。
- scc - 用于 Sun Studio C++ 编译器。
%platform% 是以下各项
- linux - 用于 Linux 系列操作系统。
- macos - 用于 Mac OS X 操作系统。
- solaris - 用于 Solaris 操作系统。
- bsd - 用于 BSD 系列操作系统。
配置是以下之一
- debug - 编译带有调试信息且无优化的库。
- release - 编译具有优化代码生成的库,并去除调试信息。
Makefiles 位于 /makefiles 目录中。
make -f icc_linux_debug all
示例:在 Linux 上使用 Intel C++ 编译 Debug 版本
gmake -f gcc_bsd_release all
示例 - 在 FreeBSD 上使用 GCC G++ 编译 Release 版本
输出文件是名为 libGeneticLibrary.a 的静态库,位于 /build/%configuration%/%compiler%_%platform% 目录中。
要将遗传算法库与应用程序链接,用户必须指定库的路径和库文件名
g++ -Wl,-L"%path_to_library%/build/%configuration%/%compiler%_%platform%"
-lGeneticLibrary -o "application_executable" [list of object files]
对于 Intel C++ 编译器,用户应该使用 icpc 命令而不是 g++,对于 Sun Studio C++ 编译器,使用 cc 命令。
%path_to_library% 是库所在目录的路径。在某些平台上,链接应用程序与遗传算法库还有其他要求。在 Linux 上,必须为链接器指定 -lrt 开关。Sun Studio 链接器需要 -library=stlport4 和 -lrt 开关,*BSD 系统上的 GNU 链接器需要 -march=i486 和 -lpthread 开关。
可移植性
要将此库移植到其他平台而对库的核心没有重大更改,目标平台必须支持
- 多线程 - 如果目标平台支持 POSIX Threads,移植会更容易,因为遗传算法库已经在类 Unix 系统上使用 Pthreads 进行多线程。
- 原子增量、减量操作以及原子比较和交换指令或原子交换操作。
- STL - 遗传算法库在某些片段中依赖于 STL 和一些非标准 STL 扩展,例如
hash_map。 - IEEE754 标准浮点数 - 库的某些部分,如随机数生成器,假定目标处理器架构支持此标准。
变更
版本 1.4
GaChromosome接口的operator==的返回值现在是bool。GaChromosome接口还添加了operator !=。此更改也影响:GaMultiValueChromosome、GaSingleValueChromosome和GaBinaryChromosome类。- 随机数生成器算法已从 RANROT 更改为 MWC。
- 方法
void GaRandomGenerator::Generate(unsigned int& word1, unsigned int& word2)的声明已更改为int GaRandomGenerator::Generate()。 - 方法
void GaRandomGenerator::Initalization(unsigned int seed)的声明已更改为void GaRandomGenerator::Initalization(unsigned int seed1, unsigned int seed1)。 enum GaRandomParams已被移除,因为它在算法更改后不再需要。struct GaState已作为GaRandomGenerator类的成员添加,它代表随机数生成器的当前状态。其成员_w和_z存储 64 位状态。- 在指定区间内生成随机数的方式已更改,以使区间内所有数字的概率相等。现在将指定给
Generate方法的最大值包含在区间内。此更改影响:GaRandomInt、GaRandomBool、GaRandomFloat和GaRandomDouble。
- 方法
GaChromosomeDomainBlock现在可以存储多个值集。已将方法const GaValueSet、* GACALL GetValueSet(int pos) const void GACALL SetValueSet(GaValueSet和* domain, int pos) int GACALL GetValueSetCount() const添加到该类。方法const T& GetClosestValue(const T& value) const的声明已更改为const T& GetClosestValue(const T& value, int pos) const。- 由
GaMultiValueChromosome类表示的多值染色体现在为每个值位置使用单独的值集。此更改也影响GaMVArithmeticChromosome类。 - 耦合操作现在可以检查生成的子代染色体是否已存在于种群中,如果存在则不将其插入结果集。该操作将此设置存储到结果集中,以便替换操作可以在将重复项插入种群之前清除它们。为此,已将
GetCheckForDuplicates和SetCheckForDuplicates方法添加到GaCouplingParams类,将SetClearDuplicates和GetClearDuplicates方法添加到GaCouplingResultSet类。此更改也影响GaMulitpleCrossoverCouplingParams。通过CheckForDuplicates函数实现检查。所有内置操作的子代染色体生成现在都在一个函数中实现:ProduceOffspring。
