实现机器学习的分步指南 XI - DBSCAN
易于实现的机器学习
引言
基于密度的噪声应用空间聚类 (DBSCAN) 是一种基于密度的聚类算法。与其他聚类算法不同,DBSCAN 将密度可达样本的最大集合视为簇。 KMeans 缺乏对非球形数据进行聚类的能力,而 DBSCAN 可以对任何形状的数据进行聚类。
DBSCAN 模型
预备知识
- ε -邻域: 对象 ε 半径范围内的对象。
- 核心点: 如果一个样本在其 ε-邻域内拥有的点数超过指定数量 (m),则该样本是一个核心点。
- 直接密度可达: 如果对象 q 位于对象 p 的 ε-邻域内,并且 p 是一个核心对象,则对象 q 从对象 p 直接密度可达。
- 密度可达: 如果存在对象链 p1,…,pn,其中 p1=p, pn=q 且对于所有 1 <= i <= n,pi+1 从 pi 直接密度可达,则对象 q 从 p 密度可达(直接密度可达和密度可达的区别在于,直接密度可达时 p 位于 q 的邻域内,而密度可达时 p 不位于 q 的邻域内)。
- 密度连接: 如果存在一个对象 o,使得 p 和 q 都从 o 密度可达,则对象 p 与对象 q 密度连接。
- 噪声: 至少不能从一个核心对象密度可达的对象。
让我们举例说明以上术语,如下图所示,其中 m = 4。由于所有红色样本的 ε-邻域内都有超过 m 个样本,因此它们都是核心点和密度可达。 B 和 C 不是核心点。 青色圆圈中的样本是直接密度可达。棕色圆圈中的样本是密度连接。 N 无法到达任何其他样本,因此它是一个噪声点。

聚类算法
首先,我们通过计算所有样本 ε-邻域内的样本数来找到所有核心点。 剩余的样本暂时标记为噪声点。 我们提供几种类型的距离,即
- 欧几里得距离
- 余弦距离
- 曼哈顿距离
def calculateDistance(self, x1, x2):
if self.distance_type == "Euclidean":
d = np.sqrt(np.sum(np.power(x1 - x2, 2), axis=1))
#d = np.sqrt(np.sum(np.power(x1 - x2, 2)))
elif self.distance_type == "Cosine":
d = np.dot(x1, x2)/(np.linalg.norm(x1)*np.linalg.norm(x2))
elif self.distance_type == "Manhattan":
d = np.sum(x1 - x2)
else:
print("Error Type!")
sys.exit()
return d
对于给定的样本,如果该样本与另一个样本之间的距离小于 ε,则它们位于同一个邻域内。
def getCenters(self, train_data):
neighbor = {}
for i in range(len(train_data)):
distance = self.calculateDistance(train_data[i], train_data)
index = np.where(distance <= self.eps)[0]
if len(index) > self.m:
neighbor[i] = index
return neighbor
然后,我们随机选择一个未访问的核心点 P 来合并与其密度连接的样本。 接下来,我们访问 P 的 ε-邻域内的样本 Q。如果 Q 是一个核心点,则 Q 与 P 属于同一个簇,并在 Q 上执行相同的过程(如深度优先搜索); 否则,访问下一个样本。 具体来说,在 P 的 ε-邻域中的搜索过程如下所示
- 选择核心点 A 并找到其 ε-邻域中的所有样本。 A 的 ε-邻域内的样本属于 A 的簇。 然后依次访问 A 的 ε-邻域内的样本。

- 访问 A 的 ε-邻域内的样本 B。 B 是一个核心样本,访问 B 的 ε-邻域内的样本。

- 访问 B 的 ε-邻域内的样本 C。 C 属于 A 并且 C 不是一个核心点。 访问 B 的 ε-邻域内的其他样本。

- 访问 B 的 ε-邻域内的另一个样本 D。 D 是一个核心点。 访问 D 的 ε-邻域内的样本。

- 访问样本 E。 E 不是核心点。 现在,没有任何点可以密度可达 A。 停止对 A 的聚类。

k = 0
unvisited = list(range(sample_num)) # samples which are not visited
while len(centers) > 0:
visited = []
visited.extend(unvisited)
cores = list(centers.keys())
# choose a random cluster center
randNum = np.random.randint(0, len(cores))
core = cores[randNum]
core_neighbor = [] # samples in core's neighbor
core_neighbor.append(core)
unvisited.remove(core)
# merege the samples density-connectivity
while len(core_neighbor) > 0:
Q = core_neighbor[0]
del core_neighbor[0]
if Q in initial_centers.keys():
diff = [sample for sample in initial_centers[Q] if sample in unvisited]
core_neighbor.extend(diff)
unvisited = [sample for sample in unvisited if sample not in diff]
k += 1
label[k] = [val for val in visited if val not in unvisited]
for index in label[k]:
if index in centers.keys():
del centers[index]
参数估计
DBSCAN 算法需要 2 个参数 ε,它指定点彼此之间的距离应该有多近才能被认为是集群的一部分; 以及 m,它指定一个点应该有多少个邻居才能包含到集群中。 以 wiki 中的一个例子为例,我们计算每个点到其最近邻居的距离,该邻居位于同一分区内,如下所示,我们可以很容易地确定 ε = 22。
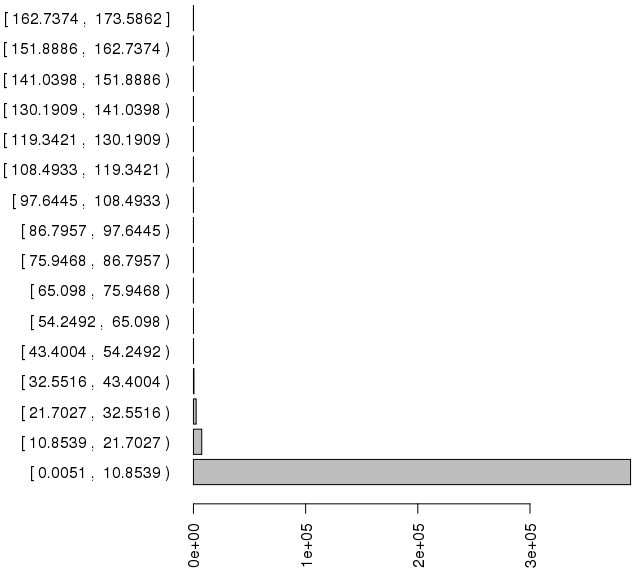
对于参数 m,我们计算核心点的 ε 邻域内有多少个样本,如下所示。 我们选择 m = 129,因为它是第一个谷底。

结论与分析
DBSCAN 能够对非球形数据进行聚类,但无法反映高维数据。 当密度分布不均匀时,聚类性能不是很好。 KMeans 和 DBSCAN 之间的聚类性能如下所示。 很容易发现 DBSCAN 比 KMeans 具有更好的聚类性能。




本文相关的代码和数据集可以在 MachineLearning 中找到。
